http://www.chemistrymag.org/cji/2004/066040pc.htm |
Jun. 1,
2004 Vol.6 No.6 P.40 Copyright |
Du Qishi a*, Wang Shuqing a, Wei Dongqing a, Li Aixiu
a,b(aInstitute of Bioinformatics and Drug Discovery, Tianjin Normal University; bMedical College of Chinese People's Armed Police Forces, Tianjin, 300162) Abstract In this research we calculate the correlation coefficients and analyze the correlation relationship among 20 amino acids in 4 type proteins (a, b, a/b, and a+b), total 204 proteins. Our research shows that amino acid correlations can be divided into strong positive correlation, strong negative correlation, weak correlation, and no-correlation. The correlation relationships of amino acids in 4 type proteins are quite different, and represent the protein's structure characters. We analyze the relationship between amino acid correlation and the structure of proteins and give explanation for the physicochemical origins of amino acid correlations.
Keywords Amino acids; Protein structure; Bioinformatics; Chemometrics 蛋白质中的氨基酸相关性及在结构分析中的应用 杜奇石*a,王树青a,李爱秀a,b
(a天津师范大学,生物信息学与药物开发研究所,天津,300074; b中国人民武装警察部队医学院,天津,300162)
2004年2月22日收稿; 本项目获得国家自然科学基金(20373048)和天津市科委基础科学重点项目(023618211)资助。
摘要
本文计算和分析了4种类型(a型、b型、a/b型、和a+b型)共计204个蛋白质中的20种氨基酸间的相关性。研究发现,氨基酸之间的相关性可分为强正相关、强负相关、弱相关、和不相关。作为蛋白质的建筑构件,20种氨基酸在不同类型的蛋白质中的相关性反映了这些建筑构件间的匹配规则,代表了蛋白质的结构特征。本文分析了部分氨基酸间的相关性与蛋白质结构间的联系,从物理和化学性质上解释了氨基酸相关性的起源。关键词 氨基酸;蛋白质结构;生物信息学;化学计量学
氨基酸是构造蛋白质的建筑材料,为了建造出有特定构型的蛋白质的结构单元(如a螺旋和b带)和整体结构(如球蛋白),这些建筑构件间必定有一定的匹配性。也就是说,在特定的蛋白质造型中,某一种氨基酸只能与一部分氨基酸连接,而不能与另一部分氨基酸连接,氨基酸之间存在着一定的相关性。在这个研究里我们把化学计量学[1]的相关性分析法应用于a型、b型、a/b型和a+b型蛋白质中氨基酸之间的相关性分析。氨基酸的相关性来源于氨基酸的物理和化学性质,如氨基酸侧链的亲水和疏水性、侧链的体积、氢键的给体和受体的数目等。我们进而分析部分氨基酸相关性的物理化学性质的起源,给出相关性的合理解释。
1 氨基酸相关性分析法
设N个已知蛋白质组成集合S,它是由m个子集Sx构成的并集,
每个子集x对应于一个蛋白质类,含nx个蛋白质,有N = Sx nx。每个蛋白质是20维氨基酸空间的一个向量Xx,k,或一个点,
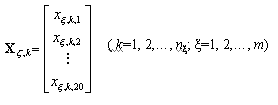 (2)
(2)式(3)中xx,k,i是子集x的第k个蛋白质的第i个氨基酸出现的百分数频率,遵守下面的归一化条件,
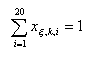 (3)
(3)每个子集Sx有一个标准向量
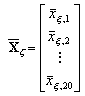 (4)
(4)它的分量
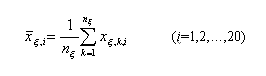 (5)
(5)子集Sx的蛋白质百分组成构成矩阵[Xx]nxx20。我们按以下公式计算子集Sx的协方差矩阵[Cx]20x20,
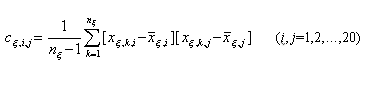 (6)
(6)协方差矩阵表达了子集内蛋白质组成对平均值的离散程度。以往在以氨基酸组成为基础的蛋白质结构预测中[2-5],着眼点是未知蛋白质的组成与标准蛋白质组成间的“距离”,未知蛋白质属于距离最小的蛋白质类型。本文提出的氨基酸相关性分析法从不同的角度考虑问题,立足点在氨基酸间的相互关联。这一方法认为在同一类型的蛋白质中各种氨基酸的数量可以有较大的变化,但氨基酸的数量变化有一定的相互制约性,遵循一定的相关关系。为此我们从协方差矩阵Cx出发,构造子集Sx的氨基酸间的相关矩阵[Rx]20x20,
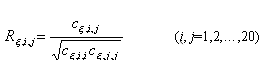 (7)
(7)相关矩阵Rx是对称矩阵,对角元均为1,Rx,i,i=1。在构成蛋白质的二级结构单元(a螺旋和b带等)时氨基酸间应存在一定的匹配规则:一种氨基酸与某些氨基酸连接的频率可能高一些,而与另一些氨基酸连接的频率低一些。相关矩阵从蛋白质的组成矩阵出发,表达了氨基酸间在数量上的依存关系,反映了蛋白质类型结构特征。
2. 计算结果
我们采用了文献[6]使用的蛋白质训练集,从数据库(PDB
Bank)中选取了4种类型共204个蛋白质(a型52个、b型61个、a/b型45个、和 a+b型46个),蛋白质的PDB代码列于表1。
表1 计算使用的204个蛋白质的PDB代码[6]
1.a型52个 |
1vls_ | 1ithA | 1spgA | 1pbxB | 1emy_ | 2pghB | |
1aep_ |
1ilk_ |
2fal_ |
2gdm_ |
1fslA |
1sctB |
1hdaB |
1hdaA |
1ash_ |
1maz_ |
2hbg_ |
2lhb_ |
1hlm_ |
1babB |
1hdsA |
1hrm_ |
1bcfA |
1mls_ |
3sdhA |
1hdsB |
1lht_ |
2asr_ |
1ibeB |
1mygA |
1cnt1 |
1rhgA |
1allA |
1myt_ |
1outA |
1babA |
1mbs_ |
1vlk_ |
1gdy_ |
1spgB |
1flp_ |
1osa_ |
1outB |
1bgc_ |
2mm1_ |
|
1hlb_ |
1sra_ |
1ibeA |
1sctA |
1pbxA |
1bgeA |
2pghA |
|
2 .b型61个 |
1yna_ | 1bbdH | 1nldH | 1bafL | 1iaiM | 1nsnH | |
1bbt2 |
3hhrC |
8fabA |
1eapA |
1opgL |
1bjmA |
1igcL |
1plgH |
1cfb_ |
6fabl |
1flrH |
1gafL |
1ospL |
1bqlH |
1ikfL |
1plgL |
1edhA |
8fabB |
1ggiH |
1gbg_ |
1vgeL |
1bqlL |
1indL |
1tetH |
1gen_ |
1pex_ |
1indH |
1ggiL |
2fbjL |
1dfbL |
1macA |
1xnd_ |
1sacA |
1vcaA |
1JELH |
1ghfH |
2mcg1 |
1forL |
1mamL |
1yuhA |
1tcrA |
1mfbL |
2cgrH |
1hilB |
7fabL |
1ghfL |
1mreH |
3hfmH |
2ayh_ |
1gnhA |
7fabH |
1ncbL |
1acyL |
1iaiL |
1ngqH |
|
3.a/b型45个 |
1nar_ | 1vdc_ | 2ebn_ | 1obr_ | 1lwiA | 1cerO | |
1amp_ |
1ghr_ |
1pbn_ |
1vpt_ |
3pga1 |
1cnv_ |
1wsaB |
1gia_ |
1ceo_ |
1gym_ |
1pfkA |
1xel_ |
8abp_ |
1exp_ |
2alr_ |
2lip_ |
1cvl_ |
1lbiA |
1sbp_ |
1xyzA |
1enp_ |
1trb_ |
3ecaA |
1ula_ |
1dorA |
1lucA |
1scuA |
2bgu_ |
1gdhA |
1ghsA |
4pfk_ |
2gbp_ |
1gca_ |
1masA |
1thtA |
2ctc_ |
1lucB |
1hdgO |
1agx_ |
|
4 .a+b型46个 |
1gtqA | 1mkaA | 1seiA | 1vhiA | 1apyB | 2prd_ | |
2aak_ |
1def_ |
1hjrA |
1msc_ |
1sfe_ |
1vsd_ |
1div_ |
1hup_ |
1afb1 |
1doi_ |
1htp_ |
1nhkL |
1snc_ |
1whtB |
1pvuA |
1nueA |
1bplA |
1epaB |
1ino_ |
1pkp_ |
1std_ |
1ytbB |
1npk_ |
1cdwA |
1cof_ |
1fil_ |
1itg_ |
1poc_ |
1tfe_ |
2tbd_ |
1qcqA |
1pne_ |
1cyw_ |
1grj_ |
1lit_ |
1rbu_ |
1vhh_ |
8atcB |
1ril_ |
2kmb1 |
根据公式(
4)和(5)计算各子集的标准蛋白质,结果示于图1。a型和b型的标准蛋白质的组成有较大差异,但a/b型和a+b型的组成十分相似。这一结果说明,仅仅依靠氨基酸的组成区分a/b型和a+b型的蛋白质有较大的难度。表2 a型蛋白质的20个氨基酸间的相关系数,根据表1中的数据计算
| A | 1 |
||||||||||||||||||
| C | -0.300 |
1 |
|||||||||||||||||
| D | -0.281 |
-0.011 |
1 |
||||||||||||||||
| E | -0.565 |
-0.016 |
0.215 |
1 |
|||||||||||||||
| F | 0.126 |
0.091 |
0.174 |
-0.132 |
1 |
||||||||||||||
| G | 0.216 |
-0.162 |
-0.140 |
-0.182 |
0.035 |
1 |
|||||||||||||
| H | -0.194 |
-0.181 |
-0.130 |
0.022 |
0.019 |
0.164 |
1 |
||||||||||||
| I | -0.269 |
-0.130 |
0.158 |
0.369 |
-0.390 |
0.036 |
-0.249 |
1 |
|||||||||||
| K | -0.036 |
-0.316 |
0.182 |
0.141 |
0.314 |
0.151 |
0.402 |
0.145 |
1 |
||||||||||
| L | -0.276 |
0.446 |
-0.214 |
-0.087 |
-0.101 |
-0.049 |
0.340 |
-0.413 |
-0.208 |
1 |
|||||||||
| M | -0.202 |
0.013 |
0.178 |
0.167 |
-0.219 |
-0.138 |
-0.583 |
0.326 |
-0.377 |
-0.278 |
1 |
||||||||
| N | 0.027 |
-0.046 |
-0.015 |
-0.064 |
0.073 |
-0.380 |
-0.241 |
-0.182 |
-0.190 |
-0.138 |
0.165 |
1 |
|||||||
| P | -0.085 |
0.465 |
-0.421 |
-0.217 |
-0.065 |
-0.044 |
0.101 |
-0.127 |
-0.341 |
0.402 |
-0.147 |
-0.318 |
1 |
||||||
| Q | -0.056 |
0.242 |
-0.487 |
0.110 |
-0.359 |
-0.258 |
-0.248 |
-0.026 |
-0.506 |
0.310 |
0.286 |
0.233 |
0.175 |
1 |
|||||
| R | -0.541 |
0.368 |
0.265 |
0.244 |
-0.218 |
-0.361 |
-0.363 |
0.185 |
-0.456 |
0.029 |
0.540 |
0.158 |
-0.036 |
0.195 |
1 |
||||
| S | 0.498 |
-0.173 |
-0.195 |
-0.436 |
-0.054 |
-0.122 |
-0.210 |
-0.233 |
-0.314 |
-0.077 |
0.079 |
-0.086 |
0.118 |
-0.034 |
-0.058 |
1 |
|||
| T | 0.037 |
-0.097 |
-0.074 |
-0.091 |
-0.240 |
-0.416 |
-0.013 |
-0.013 |
-0.274 |
-0.121 |
0.103 |
-0.036 |
0.246 |
0.157 |
0.080 |
0.167 |
1 |
||
| V | 0.277 |
-0.226 |
0.075 |
-0.421 |
0.246 |
0.235 |
0.067 |
-0.310 |
0.105 |
-0.203 |
-0.392 |
0.070 |
-0.099 |
-0.514 |
-0.273 |
0.055 |
-0.114 |
1 |
|
| W | 0.060 |
-0.189 |
-0.130 |
0.021 |
-0.004 |
0.134 |
0.055 |
0.187 |
0.013 |
-0.416 |
-0.102 |
-0.002 |
0.067 |
-0.094 |
-0.160 |
-0.046 |
-0.036 |
0.232 |
1 |
| Y | -0.297 |
0.186 |
0.262 |
0.031 |
-0.264 |
-0.288 |
-0.298 |
0.176 |
-0.136 |
-0.110 |
0.394 |
0.139 |
-0.147 |
0.022 |
0.569 |
-0.001 |
0.100 |
-0.200 |
-0.141 |
A |
C | D | E | F | G | H | I | K | L | M | N | P | Q | R | S | T | V | W |
表3 b型蛋白质的20个氨基酸间的相关系数,根据表1中的数据计算
| A | 1 |
||||||||||||||||||
| C | -0.051 |
1 |
|||||||||||||||||
| D | -0.275 |
-0.256 |
1 |
||||||||||||||||
| E | 0.021 |
-0.057 |
0.258 |
1 |
|||||||||||||||
| F | -0.178 |
-0.489 |
0.484 |
0.204 |
1 |
||||||||||||||
| G | 0.017 |
-0.447 |
-0.274 |
-0.397 |
0.031 |
1 |
|||||||||||||
| H | -0.071 |
-0.138 |
0.022 |
0.273 |
0.122 |
0.104 |
1 |
||||||||||||
| I | -0.291 |
-0.387 |
0.660 |
0.433 |
0.492 |
-0.281 |
0.114 |
1 |
|||||||||||
| K | 0.010 |
0.098 |
0.227 |
0.272 |
0.373 |
-0.334 |
-0.210 |
0.223 |
1 |
||||||||||
| L | -0.213 |
0.350 |
-0.099 |
0.030 |
0.027 |
-0.086 |
-0.078 |
-0.218 |
0.191 |
1 |
|||||||||
| M | -0.226 |
-0.083 |
0.037 |
0.109 |
0.119 |
-0.175 |
0.021 |
0.003 |
0.134 |
-0.258 |
1 |
||||||||
| N | -0.155 |
-0.593 |
0.328 |
0.029 |
0.366 |
0.268 |
0.287 |
0.413 |
-0.255 |
-0.599 |
0.152 |
1 |
|||||||
| P | 0.373 |
0.103 |
-0.154 |
-0.017 |
-0.130 |
-0.304 |
-0.140 |
-0.159 |
0.208 |
0.214 |
-0.055 |
-0.466 |
1 |
||||||
| Q | 0.133 |
0.307 |
0.027 |
-0.099 |
-0.184 |
-0.456 |
-0.277 |
0.027 |
-0.054 |
-0.006 |
-0.291 |
-0.149 |
-0.065 |
1 |
|||||
| R | -0.378 |
0.014 |
0.448 |
0.292 |
0.193 |
-0.255 |
0.140 |
0.451 |
-0.168 |
0.177 |
-0.036 |
0.079 |
-0.223 |
0.076 |
1 |
||||
| S | -0.049 |
0.608 |
-0.329 |
-0.445 |
-0.506 |
-0.210 |
-0.465 |
-0.321 |
-0.051 |
0.115 |
-0.169 |
-0.431 |
-0.049 |
0.455 |
-0.103 |
1 |
|||
| T | 0.065 |
0.380 |
-0.482 |
-0.321 |
-0.566 |
0.116 |
-0.023 |
-0.568 |
-0.325 |
-0.006 |
0.086 |
-0.220 |
-0.054 |
0.030 |
-0.327 |
0.358 |
1 |
||
| V | 0.300 |
-0.007 |
-0.466 |
-0.156 |
-0.281 |
0.147 |
0.033 |
-0.543 |
-0.276 |
0.196 |
-0.015 |
-0.363 |
0.493 |
-0.181 |
-0.263 |
-0.111 |
0.198 |
1 |
|
| W | -0.042 |
-0.432 |
-0.126 |
-0.205 |
0.157 |
0.551 |
0.113 |
-0.058 |
-0.164 |
-0.290 |
0.139 |
0.328 |
0.015 |
-0.475 |
-0.352 |
-0.428 |
-0.069 |
0.042 |
1 |
| Y | -0.112 |
-0.475 |
-0.014 |
-0.412 |
0.032 |
0.609 |
-0.016 |
-0.081 |
-0.160 |
-0.362 |
0.117 |
0.402 |
-0.376 |
-0.230 |
-0.218 |
-0.119 |
-0.109 |
-0.138 |
0.498 |
A |
C | D | E | F | G | H | I | K | L | M | N | P | Q | R | S | T | V | W |
我们按公式(6)和(7)计算相关矩阵,由于对称关系,我们仅在表2列出了a型蛋白质的相关矩阵的下三角形部分。表3是b型蛋白质的相关矩阵。20个氨基酸之间共有C220=190个氨基酸相关对。相关系数应在0到1之间,代表了从完全不相关到完全相关。表1和表2中的大多数相关系数都远小于1,说明了多数氨基酸间没有特定的相关性,有较大的可替换性。但也有个别氨基酸对的相关系数明显高于平均值。根据氨基酸对的相关系数的大小,我们把氨基酸相关性分为强正相关(相关系数>0.5)、强负相关(相关系数<-0.5)、弱相关(|相关系数|<0.05)、和不相关(|相关系数|<0.01)。我们把a型蛋白质和b型蛋白质中几个有强正相关和强负相关的氨基酸对的相关关系示于图2、图3、图4和图5。




图1 4种类型蛋白质(a型52个、b型61个、a/b型45个、和a+b型46个)的氨基酸平均含量。a型和b型有较大差异,但a/b型和a+b型的组成十分相似

图2 a型蛋白质中氨基酸H与M间的相关关系,相关系数RH-M=
-0.583。氨基酸H与M为负相关,H的含量高时,M的含量低


图4 b型蛋白质中氨基酸D与I间的相关关系,相关系数RD-I=0.660。氨基酸D与I为正相关,D的含量高时,I的含量也高

图5 b型蛋白质中氨基酸V与I间的相关关系,相关系数RV-I=-0.543,氨基酸V与I为负相关,V的含量高时,I的含量反而低
3. 结果分析
在a型蛋白质中最大的相关系数在氨基酸M与H间,RM-H=
-0.583,说明氨基酸M与H间有一定的制约关系,表现为强的负相关(图2)。值得注意的是在a型蛋白质中氨基酸M和H的出现频率都不很高,参看图1。在b型蛋白质中最大的相关系数在氨基酸D与I间,RD-I=0.660,说明氨基酸D与I间有强的正相关(图4)。



图6 天冬氨酸(D, Asp)、异亮氨酸(I, Ile)和缬氨酸(V, Val)的分子结构式和侧链的性质
氨基酸间的相关性反映了氨基酸之间的制约关系,下面我们以b型蛋白质艾滋病毒的HIV-1 GP120 蛋白质(1acyL)为例,说明在b型蛋白质中天冬氨酸(D, Asp)和异亮氨酸(I, Ile)间的强正相关关系的物理化学起源。图6给出了D(天冬氨酸Asp)、I(异亮氨酸Ile)、V(缬氨酸Val)三种氨基酸的结构式。天冬氨酸D有强亲水性侧链CH2COO-,异亮氨酸I的侧链和缬氨酸V的侧链都是强疏水性的。
1 DIVMTQSPAS LVVSLGQRAT ISCRASESVD SYGKSFMHWY QQKPGQPPKVEEEE SE EEEETT EE EEEEESS E ETTEE EEEE EE TTS EE
51 LIYIASNLES GVPARFSGSG SRTDFTLTID PVEADDAATY YCQQNNEDPP
EEGGGEE T TTTTTEEEEE ETTEEEEEES S GGG EEE EEEE SSSS
101 TFGAGTKLEM RRADAAPTVS IFPPSSEQLT SGGASVVCFL NNFYPKDINV
EE EEEEE E B EEE EE HHHHH TTEEEEEEEE ESEESS EE
151 KWKIDGSERQ NGVLNSWTDQ DSKDSTYSMS STLTLTKDEY ERHNSYTCEA
EEEETTEE SSEEEEE TTT B EE EEEEEEHHHH HT SEEEEEE
201 THKTSTSPIV KSFNR
EETTBSS EE EEEE图7 艾滋病毒的HIV-1 GP120 蛋白质1acyL的氨基酸序列和二级结构解析[7,8]
图7是艾滋病毒的HIV-1 GP120 蛋白质(1acyL)的氨基酸序列和二级结构解析[7,8]。在图7中我们找到了4个氨基酸D和I紧密相连处,在图中用黑体字母标出。有趣的是这4处都出现在b折叠带的头或尾的部位,也就是b带发生折叠的地方。氨基酸的疏水和亲水特性在蛋白质的结构中起十分重要的作用[9,10]。Rose的研究表明[11,12],蛋白质的氨基酸的疏水特性和序列的疏水区域是关联的,并基于此来判断二级结构单元的拐角。由于D是强亲水性氨基酸而I是强疏水性氨基酸,这就是D与I经常成对出现在β带拐角处的原因,由此造成了D与I的强正相关。氨基酸D与I除成对出现外还在多处分散出现,属于随机加相关分布,因而整体系相关系数不是很高(0.660)。图5显示在b型蛋白质中V(缬氨酸Val)与I(异亮氨酸Ile)间的较强的负相关。从图6中V和I的分子结构可知二者均为强疏水性氨基酸,仅相差一个亚甲基(-CH2-)。在b型蛋白质的疏水部位,二者中有一个出现时,就会减少另一个出现的概率,这可能是V和I表现出强负相关性的原因。
4.结论
理论分析和计算实践都表明在各种类型的蛋白质中,氨基酸之间存在着特定的相关性。氨基酸间的相关性起源于氨基酸的特定的物理化学性质。有一些氨基酸对的相关性比较明显,容易作出解释,如b型蛋白质中的天冬氨酸D与异亮氨酸I间的正相关和异亮氨酸I与缬氨酸V间的负相关。但是,由于蛋白质结构的复杂性,不是所有的氨基酸对的相关性都发生在相邻的氨基酸间,像氨基酸D与I那样。相关性也有可能发生在相距比较远的氨基酸之间。这些氨基酸对的相关性的内在因素不明显,不容易给出解释,我们将在今后的工作中逐步破解。作为蛋白质的基本建筑构件,氨基酸间的相关性是这些构件的匹配规则,代表了蛋白质类型的结构特征。氨基酸相关性的研究可以用于蛋白质结构类型的预测上,我们将在以后的研究中报道。
REFERENCES
[1] Xu Lu. Chemometrics. Science Publishing House, 1985.
[2] Chou K C. Current Protein and Peptide Science, 2000,1: 171.
[3] Chou K C. Proteins: Structure, Function and Genetics, 1995, 21: 319.
[4] Chou K C. FEBS Letters, 1995, 363: 127.
[5] Chou K C, Liu W, Maggiora G M et al. Proteins: Structure, Function and Genetics, 1998,
31: 97.
[6] Berman H M, Westbrook J, Feng Z et al. Nucleic
Acids Research, 2000, 28: 235.
[7] Ghiara J B, Stura E A, Stanfiled R L et al. Science, 1994, 82: 264.
[8] http://www.rcsb.org/pdb/
[9] Qi J X, Xiao Y. Science B, 2003, 47 (6): 425.
[10] Qiu J D, Liang R P, Zou X Y et al. Chemical Journal, 2003, 61: 748.
[11] Rose G D ,Wolfonden R. Hydrogen bonding hydrophobicity packing
and protein folding, Ann Rev Biophys Biochem Struct, 1993, 22: 381.
[12] Mandell A J, Selz K A, Shlesinger M F. Physica A, 1997, 244: 254.