http://www.chemistrymag.org/cji/2006/084027rc.htm |
Apr. 2, 2006 Vol.8 No.4 P.27 Copyright |
Research progress of CTG repeats in structural biology
Hu Haiming, Xue Yonglai, Feng Xizeng
(The Key Laboratory of Bioactive Materials Ministry of Education, China, College of Life
Science, Nankai University, Tianjin, 300071)
Abstract Myotonic dystrophy
type1(MD) is an autosomal dominant neuromuscular disease, its heredity mechanism is the
expansions of CTG repeats in the 3'-untranslated region of the myotonic dystrophy protein
kinase(DMPK). This paper introduces the structure feature of CTG repeats, biochemical
feature, expansion mechanism, factors that influence its expansion, and the relationship
between expansion and MD.
Keywords Myotonic dystrophy, trinucleotide repeats CTG, expansion, structure, S-DNA
CTG重复序列的结构生物学研究进展
胡海明,薛永来,冯喜增
摘要 强直性肌营养不良(MD)是一种常染色体显性遗传神经肌肉疾病,它的遗传机制是位于肌张力蛋白激酶3’端非编码区CTG重复序列异常扩增引起的。本文概述了CTG重复序列的结构特点、生化特征、扩增机制、影响其扩增的因素以及扩增与相关疾病发生的关系等方面的最新研究进展。
关键词 强直性肌营养不良 CTG 扩增 结构 S-DNA
2006年3月7日收稿;国家自然科学基金重大研究计划-理论物理学及其交叉科学若干前沿问题(90403140)资助项目。
1. 引言
1951年,Mohr描述了DM基因和Lutheran血型基因呈连锁遗传。后来他们又发现DM和C3基因连锁,由于C3基因定位于19号染色体,因此,DM基因也被定位于19号染色体。自1992年开始,大量的研究表明:DM患者的致病基因定位于常染色体19q13.3,内有一个含G的保守序列,其编码的蛋白是一个ATP结合位点。对于三核苷酸重复序列扩增引起遗传性疾病的机制,大量的实验数据表明此重复序列的异常复制与其异常扩增有关[6]。也有的学者认为,寡核苷酸重复序列扩增的致病机理可能有以下几种方式:(1)寡核苷酸串连重复序列扩增影响它所在的基因的表达;(2)寡核苷酸重复序列扩增影响它所在的邻近基因的表达;(3)导致转录的mRNA在细胞核内持续累积,结合吸附而耗去一些影响细胞基本活动的重要蛋白,扰乱了细胞的生物化学功能,从而引起广泛症状[7-10]。因此本文概述了CTG重复序列的结构特点、生化特征、扩增机制、影响其扩增的因素等方面的内容。
2.CTG的结构特点
生物物理和生物化学技术揭示了CTG重复序列在DNA复制过程中通过碱基错配能够形成稳定的发夹结构,导致这种稳定结构形成的原因还不是很清楚。并且也有学说认为碱基间的非正常配对可能是DNA突变的起因,因此对这种非正常配对结构的认识就显得相当有必要。
在DNA复制、修复、重组过程中经常伴有单链CTG重复序列的滑动,这样就容易形成T·T配对(如图2所示),并且这些 T·T配对仍然保留在螺旋内部[11]。而在正常的Watson-Crick 碱基对中是A·T、G·C配对(如图1所示)。这种非正常的碱基配对在世代传递中就很可能引发基因突变,对后代产生一定的危害。
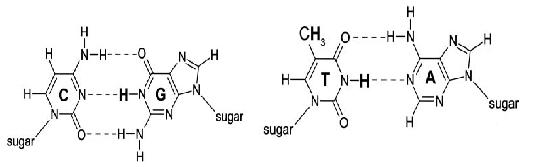
图1 Watson-Crick 配对
Fig. 1 Watson-Crick match
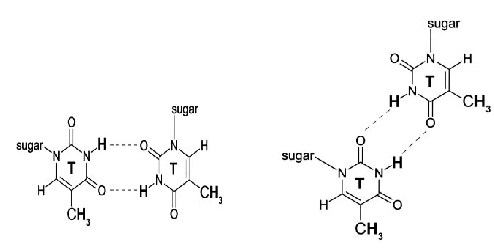
图2 T·T错配
Fig. 2 T·T mismatch 这些非正常的碱基配对有助于重复序列形成一些特殊的二级结构,而这些形成的高级结构很可能对重复序列的扩增起了一定的推动作用。
2.2 (CTG)n的结构
关于CTG重复序列的结构,迄今为止,研究比较清楚的还是重复序列数目较小的序列。Lai
Man Chi 和Sik Lok Lam等应用核磁共振(NMR)技术对含1-10个重复序列的结构
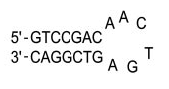 |
 |
| 图3 环内没有氢键的发夹结构
Fig. 3 Hairpin without intra-loop |
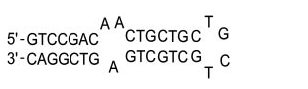
图4
环内包含四个核苷酸的发夹结构 |
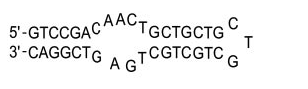
图5 环内包含三个核苷酸的发夹结构
Fig. 5 Hairpin
with a three-nucleotide loop
尽管热力学实验表明构成发夹结构的环为4-5个碱基时,相对较为稳定,且TGCT环以5'-C
3'-G碱基对封闭环,这也更有利于发夹结构的稳定;而(CTG)5
、(CTG)7 、(CTG)9形成环却只有三个碱基,并且以5'-G
3'-C碱基对封闭此环,而此结构并不怎么有利于环的稳定,因此推测它可能需要更多的Waston-Crick碱基对来维持这种发夹结构的稳定性。
他们还发现在含有小于10个CTG重复序列的结构中,若含有偶数个重复序列,相对来说更容易形成发夹结构,这点可以从(CTG)4、(CTG)6、
(CTG)8、 (CTG)10 他们所形成的发夹结构的数目高于(CTG)5
、(CTG)7 、(CTG)9来说明(如图6所示)。
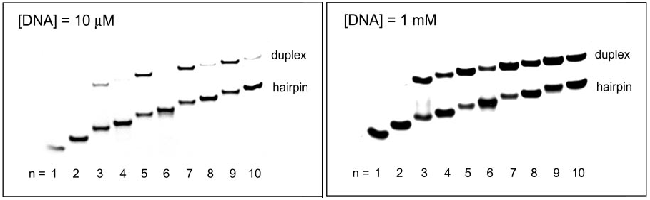
图6 Lai Man Chi and Sik Lok Lam的非变性凝胶电泳实验,DNA浓度分别为10
mΜ和1mM。
Fig.6 Non-denaturing gel of
(CTG)n at 10 mΜ and 1mM,
Lai Man Chi and Sik Lok Lam.
在小于10个重复序列的结构中,含有偶数个重复序列结构形成的是TGCT环[9]。大于10个重复序列的奇数的(CTG)15 和(CTG)25构成的也能形成TGCT环,而(CTG)16和(CTG)20来说形成的是CTG环
[12]。所以,重复数目和结构之间的关系并不是很确定,并且含有不同的重复数目也可能形成相同的发夹结构。当然除了形成发夹结构以外还包括上面所涉及的二聚体结构。二聚体结构之间也包括了非Waston-Crick碱基对。
2.3 CTG与S-DNA
含有非Waston-Crick配对的序列特异性DNA二级结构可能是诱导基因突变的一种重要的媒介。三核苷酸重复序列本身就能够形成这种不稳定的结构--滑动DNA链。一般来说包含这种重复序列的滑动DNA链有两种:S-DNA和SI-DNA。S-DNA的一条链含有CTG重复序列,而另一条互补链则含有等量的CAG重复序列,他们之间形成稳定的Waston-Crick配对。而SI-DNA的两条链所含的CTG和CAG重复序列数目并不相同[13]。有研究指出S-DNA并不一定维持在超螺旋状态,但它们在生理盐浓度条件下是很稳定的。S-DNA能够使DNA复制、修复、重组或者转录过程出错,造成基因突变[13,14]。
S-DNA结构的相对稳定性可能依赖于重复单位的序列特异性和重复数目的大小。如果重复序列长度增加,必将增加S-DNA异构体的复杂性,使其稳定性受到影响。如果在重复序列中加入一些干扰序列,就不容易形成这种滑动结构。很可能是因为S-DNA和SI-DNA并不像其它一些特异的结构(比如十字架结构和Z-DNA),它们并不是维持在超螺旋状态。
3.CTG的生化特征
与DM有关的CTG重复序列在正常人群中的重复数一般为3~35,且遵循孟德尔遗传规律,在世代传递中很少发生序列变化。而当重复序列的数目在50~80时,就会在世代交替中出现频繁的扩增现象, CTG重复数增加至200左右。假如重复数超过80时,这一序列的不稳定性明显增加,导致更高频率的跳跃式扩增,扩增后的重复数为120~1250左右[15]。这种不稳定性与CTG重复数的非线性关系或许可以这样来解释,当CTG重复数<80时,CTG重复序列发生相对较小的变化,而当此重复序列超过80时,经历一个不稳定的复制过程,在此过程中大量扩增片段,并且概率也不断的增加。
在原核生物中,当CTG重复序列数n<Okazaki fragment(冈崎片段)时,主要是增加式扩增,而当n> Okazaki fragment(冈崎片段)时,跳跃式扩增增多。Partha S. Sarkar和Haw-Chin Chang, F等研究表明,在细菌中(CTG)120~500这一范围内,CTG的扩增率增加了将近12倍[16]。由此可见,当CTG重复数增加的时候扩增的数目也增加,并且扩增的概率也大大提高了。推测其后代的扩增数目也将进一步增大,这样可以来解释为什么DM家族的病人,后代发病年龄提前的问题。
CTG重复序列遗传变异特点的表现过程主要是与转录相关[2],Marzena Wojciechowska等指出由转录过程中形成的变异片段跟单纯的复制过程中形成的相比,其变异率更大。他们认为片段切除是依赖于修复反应,并且被转录过程所放大。
3.2 CTG扩增的调节
依赖于序列长度的扩增与其形成的非正常的二级结构有关,这些非正常的二级结构能够聚合形成复合物,使得模板链和新合成的链之间不能够正常配对,因而导致序列扩增现象[6]。在DNA双链中,那些含有重复片段的非B-DNA结构很容易造成链的断裂,单链或者双链皆有可能[16]。这些断裂的区域可以由重组蛋白A(RecA)依赖的同源重组反应所修复[17]。CTG的扩增需要在SbcC缺失的情况下才能够进行,John C. Connelly和David R. F等人的实验证明SbcC的突变能够促进发夹结构的形成[18]。SbcC和SbcD是构成ATP依赖的双链核苷酸外切酶(SbcCD)活性的有效成分。其中SbcD表现SbcCD中非单链核苷酸内切酶活性,但这种酶活性的表现并不依赖于ATP,这也表明了SbcD包含在SbcCD酶活力的催化中心。 SbcC则调节SbcCD的活力。当SbcC缺失的情况下,就容易在冈崎片段中形成一些特殊的二级结构(比如ss区,或者并不完整的ds区),而这些片段就为CTG的扩增奠定了基础。
单链DNA结合蛋白(SSB)是DNA复制、修复和重组中很重要的因子,因为SSB能够阻止DNA二级结构的形成[19]。在DNA二级结构位点,复制过程会暂时停止,因此在这些位点就比较容易发生移码突变。SSB就能够抑制DNA二级结构的形成,使复制正常进行。大肠杆菌研究实验中表明SSB对维持三核苷酸重复序列的稳定起重要作用,若大肠杆菌含有温度敏感的SSB突变体,(CTG)n重复序列在42℃的稳定性与在32℃培养的细胞或者42℃的ssb+细胞相比下降。含有CTG重复序列的单链DNA构成的发夹结构很不稳定,也可能导致DNA聚合过程中错误率的增加。发夹结构能够暂时停止DNA聚合酶的作用,而这些暂时终止位点都有很高的变异率。SSB通过阻止DNA二级结构的形成来稳定三核苷酸重复序列。因为发夹结构或其它一些DNA二级结构是导致一些与人类遗传疾病相关的核苷酸重复序列稳定性的重要影响因子。
在细菌、酵母和哺乳动物细胞中三核苷酸重复序列的稳定性还依赖于复制起始点的方向性,长片段重复序列如果位于滞后链上,更倾向于序列切除,相反当编码序列位于前导链上则更倾向于序列扩增,即使概率很低[2,6,20]。推测由这些重复序列的构成的非正常的二级结构不管是在滞后链上还是新合成的链上都各自削落了DNA的复制、丢失或者是扩增。在正常人体中,这些重复序列被一些其它不相关的区域所打断,在酵母中也有干扰序列使这些三核苷酸重复序列的稳定性增加,尤其是当这些干扰序列位于(CTG)·(CAG)的5'端[6]。
如果说DNA的二级结构是重复序列扩增中的限速步骤的话,那么复制时的温度可以说是调节这过程能否发生[20]。因为复制时的温度既可影响这种二级结构的形成,也影响其结构的稳定性。Partha S. Sarkar等研究表明pTV-[CTG]140 和pTV-[CTG]200在37℃时,发生大频率的片段切除。相反在25℃时,质粒表现出明显的扩增趋势。在电泳图谱上显示出离散型的条带,分布范围[CTG]330 to .[CTG]2000 。除了受到温度的影响外,CTG的扩增就同时还受到复制时的方向的影响。当冈崎片段编码重复序列,并且复制温度在16℃-25℃时,扩增占优势地位,当滞后链编码CTG重复序列,且温度在37℃时,切除占优势地位。Partha S. Sarkar等人就提出如果复制时得温度比较低而且冈崎片段编码CTG重复序列的话就倾向于扩增表达,相反如果温度过高,且滞后链编码重复序列的话,就倾向于序列的切除。
当然序列扩增还受到DNA复制事件的影响,如果复制事件发生的次数越多,序列扩增就越多。因此如果要获得大量扩增序列的话,就得大量培养含有重复序列的质粒DNA转染的细胞。
3.3 CTG的特征常数
(CTG)n的熔解温度基本不依赖于重复序列的长度,Samir Amrane等人通过紫外吸收实验测得CTG寡核苷酸序列的熔解度,当n为6时熔解度是54℃,而当n为25时熔解度为58.7℃。但这种三核苷酸重复序列的热稳定性与KCI的浓度有一定的关系,研究发现(CTG)8寡聚核苷酸的Tm值的确依赖于KCI的浓度,当 KCI浓度增加10倍的时候,熔解度增加了7℃(10mM KCI为50℃,当100mM KCI时,熔解度为57℃)这种对离子强度的依赖性比之前报道的有所降低,且比规则的双链发夹结构低。
3.4 CTG重复序列与P53的作用
K Walter等人[21]研究发现P53能够选择性的结合到CTG重复序列的某些特定的区域,这些区域一般是形成发夹结构的茎干上,当 P53选择性的结合到这些区域的时候,使得其免于DNaseI的水解,从而起到一定的保护作用。这种结合方式与DNA立体结构特异性识别相一致,在立体结构识别中DNA的空间结构决定了P53的结合位点,因此可以推测P53结合到(CTG)n发夹结构的位点特异区是由DNA的三维结构决定的。P53结合到(CTG)·(CAG)区依赖于DNA的构象, P53相对来说更容易结合到由CTG或者CAG构成的发夹结构,当P53浓度比较低时,与P53结合的两个区域接近与DNA的末端,这两个结合区各包含了3个CTG重复序列,并且被其它两组裸露的重复序列所分离。当然P53也可以结合到(CTG)·(CAG)构成的线性B-DNA或者错配的二聚体结构上面。结合到错配二聚体结构上以后能更进一步的诱导DNA拓扑结构的变化,他们还发现随着P53浓度的增加,与其结合的重复序列变得对DnaseI非常敏感,而DnaseI的敏感区依赖于机构的变化,因此推测P53很可能是DNA拓扑结构的调节因子。
4.CTG研究展望
[1] E R Moxon, C Wills. DNA microsatellites: Agents of evolution Sci A m, 1999, 280, 94-100.
[2] M Wojciechowska, A Bacolla, J E Larson et al. J. Biol. Chem., 2005,280, 941-965.
[3] Zhao X P. Guowai YIxue (shenjinbinxue Shenjinwaike) , 2004, 31: 134-137.
[4] C A Thornton, J P Wymer, Z Simmons, et al. Nature Genet, 1997,16, 407-409.
[5] A V Philips, L T Timchenko, T A Cooper, Science, 1998, 280, 737-741.
[6] R Pelletier, M M. Krasilnikova, G M. Samadashwily, et al. Mol. Cell. Biol., 2003, 23, 1349-1357.
[7] K Ricker, T Grimm, MC Koch, et al. Neurology, 1999, 52, 170-171.
[8] J Finsterer. Eur J Neurol , 2002, 9, 441-447.
[9] J W Day, K Ricker, J F Jacobsen, et al. Neurology, 2003,60, 657-664.
[10] Yu Z L, Xie H J, Niu F et al. Zhonghua Shenjin YIxue Zazhi, 2005 , 4: 225-228.
[11] L M Chi, S L Lam, Nucleic Acids Res., 2005, 33, 1604-1617.
[12] M Tam, S E Montgomery, M Kekis, et al. J. Mol. Biol. 2003, 332, 585–600.
[13] C E Pearson, Y H Wang, J D Griffith, et al. Nucleic Acids Res., 1998, 26, 816-823.
[14] C E Pearson, R R Sinden, Biochemistry, 1996, 35, 5041–5053.
[15] S Amrane, B Sacca, M Mills, et al. Nucleic Acids Res., 2005, 33, 4065-4077.
[16] C Jankowski, F Nasar, D K Nag, Proc. Natl. Acad. Sci. 2000, 97, 2134-2139.
[17] M L Hebert, L A Sptiz, R D Wells, J. Mol. Biol, 2004, 336, 655-672.
[18] J C Connelly, D R F Leach, Genes to Cells, 1996, 1, 285-291.
[19] W A Rosche, A Jaworski, S Kang, et al. Journal of Bacteriology, 1996,178, 5042-5044.
[20] P S Sarkar, H C Chang, F. B Boudi, et al. Cell, 1998,13, 531-540.
[21] K Walter, G Warnecke, R Bowater, et al. J. Biol. Chem., 2005, 10, 1074.