http:///www.chemistrymag.org//cji/2008/101004pc.htm |
Jan. 1,
2008 Vol.10 No.1 P.4 Copyright |
Shi Huijie, Shi Xianfa
(Chemistry Department of Tongji University, Shanghai 200092
Keywords enzyme mimic, calixarene, cleavage of DNA, molecular recognition
新型杯芳烃衍生物与DNA作用性质的研究
史慧杰,施宪法
(同济大学化学系 上海 200092)
摘要 本文采用紫外滴定和粘度测试的方法研究了新型杯芳烃衍生物UC与CT-DNA的相互作用,结果发现:UC可以以经典插入的模式与DNA作用,并与DNA形成1:1的超分子化合物;我们推测可能是UC中的碱基尿嘧啶插入到了DNA的碱基对中间,起到了识别DNA的作用。同时,我们采用琼脂糖凝胶电泳的方法,研究了pBR322 DNA的断裂情况,结果发现:在温度37
oC条件下,UC的存在对H2O2断裂pBR322 DNA起到了超分子催化的作用,使DNA从闭环超螺旋的Form I 型转变为缺刻的Form II型。关键词 模拟酶,杯芳烃,DNA断裂,分子识别
1 概要
核酸断裂与基因重组技术是分子生物学和基因工程学的核心技术,其中对DNA和RNA的定位切割又是该技术的关键。人们试图通过化学的方法合成能够定点切割DNA的分子剪刀或者酶的模型物,通过DNA的定点切割或者氧化断裂,水解断裂等,寻找储存在DNA分子中的遗传信息,了解生命的奥秘,为医治各种遗传病,肿瘤等建立理论的基础。
近年来,模拟酶的研究一直是人们关注的焦点。杯芳烃作为一种优秀的分子平台也广泛用于模拟酶的研究中。杯芳烃是一种人工合成的由取代苯酚围成的环状超分子受体化合物[1]。杯芳烃及其衍生物的分子具有大小可调的疏水空腔,可以接纳识别大小合适的小分子,同时上下沿可以进行化学修饰,引入不同的官能团作为活性位点。因此,杯芳烃及其衍生物可以同时提供疏水环境和活性位点,从而使其在生物大分子,如蛋白质,核苷及核苷酸的催化断裂中具有广泛的应用前景[2-7]。目前,杯芳烃衍生物的模拟酶研究主要是受人体内酶的结构的启发,在杯芳烃的骨架上引入各种金属配合物分子,使其作为催化反应的活性位点,进而研究其对DNA或者其模型物的催化水解作用等等[8-11]。杯芳烃本身由于缺少识别基团,不能够识别指定的核苷酸或者DNA分子,从而限制了它们DNA和核苷酸定点切割中的应用,因此本文作者尝试将有机小分子引入到杯芳烃的骨架当中,使其能够识别DNA分子,进而催化DNA的断裂。这方面的工作还很少有报道[12]。
作者采用分子设计的方法,以杯芳烃为分子平台,以尿嘧啶作为潜在“识别子”,设计合成了含有尿嘧啶基团的衍生物UC(5-(Uracil-N1-acetamido)-25, 26, 27, 28-tetrahydroxycalix[4]arene,即:5-(尿嘧啶-N1-乙酰氨基)-25, 26, 27, 28-4-羟基杯[4]芳烃),并通过质谱和LB膜的方法研究了UC对核苷和碱基的选择性识别,结果证明UC可以从多种混合的碱基和核苷的溶液中,选择性的识别出腺嘌呤和腺苷(合成路线及分子结构见图1)。这部分的工作已经报道过[13]。在本文中,我们采用紫外和粘度的方法研究了UC与DNA的作用,并采用琼脂糖凝胶电泳的方法对UC催化pBR322 DNA的断裂作用进行了初步探索,希望能够为开发出一种新型的能够定点切割DNA的模拟酶试剂奠定一定的基础。
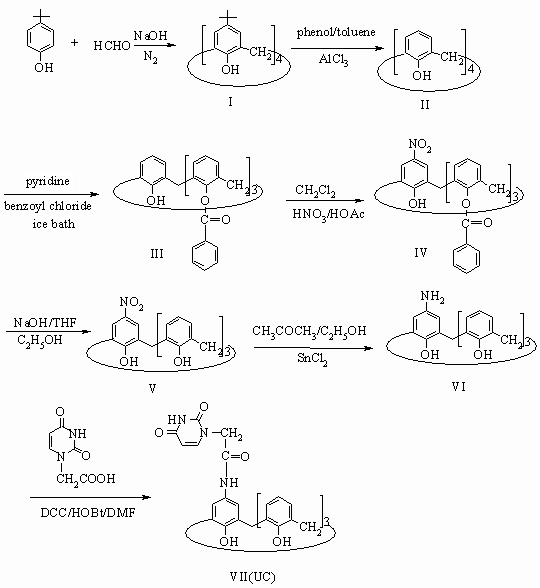
图1 UC的合成路线及分子结构图
Fig.1 The structure and synthesis route of UC
2 实验部分
2.1 仪器及试剂
Lambda Bio 40 UV/VIS型 紫外可见光谱仪(美国PE公司);乌氏粘度计(毛细管内径0.57mm,上海申立玻璃仪器有限公司);DYY-11型电泳仪(北京六一仪器厂);JY02G型凝胶成像仪(北京君意东方电泳设备有限公司);小牛胸腺DNA(CT-DNA)购自sigma公司,pBR322 DNA购自Fermentas公司。
其他所有使用的试剂均为市售分析纯或生物试剂,未经注明均未进行处理。
2.2 溶液的配置
2.2.1 缓冲液的配置
所有溶液的配置均使用Tris缓冲液。
Tris缓冲液1:含有5 ´ 10-3mol× L-1 Tris和0.05mol× L-1 NaCl,用盐酸调节酸度使pH = 7.2。UC与DNA的相互作用的紫外和粘度实验均在此缓冲溶液中进行。UC先用不超过10%的DMSO溶解,再用此Tirs缓冲液定容。
Tris缓冲溶液2. 含有0.05 mol× L-1Tris,0.018 mol× L-1 NaCl,用盐酸调节酸度使溶液pH = 7.2。此电泳缓冲液只适用于电泳反应液的配制。
TBE电泳缓冲液的配置 首先将10.8 g Tris,5.5 g H3BO3和0.9 g EDTA溶解于1000 mL水中,配成 TBE的硼酸溶液,浓度为0.089 mol× L-1 Tris,0.089 mol× L-1 H3BO3 和2 ´ 10-3 mol× L-1 EDTA(pH = 8.3)。
2.2.2 UC溶液的配置
精确称量一定量的UC,用Tris缓冲液1配置成浓度为4´ 10-5 mol× L-1的溶液,用于紫外和粘度的研究实验。
精确称量一定量的UC,用Tris缓冲液2配置成浓度为1´ 10-4 mol× L-1的溶液,用于琼脂糖凝胶电泳的研究实验。
2.2.3 DNA溶液的配置及浓度的确定
称取适量的小牛胸腺DNA溶于Tris缓冲溶液 (pH =7.2)中,抽滤,将所得到的滤液按需要稀释至一定浓度。测量DNA溶液在260 nm和280 nm处的吸光度值,如果A260 / A280 = 1.8-1.9,说明基本上不含蛋白质,不需要再进一步处理。小牛胸腺DNA的浓度以碱基对的摩尔浓度计,测量DNA在260 nm处的吸光度,然后根据DNA在260 nm处的摩尔吸光系数6600 L× mol-1cm-1来确定DNA的浓度。
[DNA]=K× A260/6600
K为稀释的倍数。测量DNA吸光度时,如DNA浓度过高,可导致吸光度测量不准,一般应稀释到A在0.5-1之间较为合适。配制好的DNA储备液应在4 oC温度下保存,并于4天内使用。
2.2.4 DNA粘度的测定
DNA溶液的粘度用乌氏粘度计测定,测定时将溶液的温度恒定在30 ± 0.1oC。保持DNA浓度不变([DNA]=2.5´ 10-4mol× L-1),改变[UC]/[DNA]的比例([UC]/[DNA]= 0.03,0.06,0.09,0.12),测得不同UC浓度下DNA的相对粘度。测试液的相对粘度按公式h = (t – t0) / t0计算,其中t0为缓冲液流经毛细管所需的时间,t为DNA溶液(含浓度不等的UC)流经毛细管所需的时间。以(h / h0)1/3对结合比率r (r = [UC] / [DNA])作图。h0为未加UC时DNA溶液的相对粘度。
2.2.5 琼脂糖凝胶电泳实验
0.8%的琼脂糖凝胶电泳,pH = 8.3的Tris-H3BO3-EDTA缓冲溶液作为电极缓冲溶液,溴酚蓝溶液作为电泳过程中的指示标志,向含有pBR 322 DNA 的溶液中加入一系列不同体积的化合物UC溶液,混合均匀,使反应液的终体积保持在10 mL。将制备好的电泳反应液在37° C恒温12小时,然后将此电泳反应液置于TBE电泳缓冲液中,在60V,60mA的恒压恒流条件下电泳一段时间后,以溴化乙锭为显色剂,最后用JY02G型凝胶成像仪分析结果拍摄照片。
3 实验结果及讨论
3.1 紫外光谱法研究UC与CT-DNA
紫外光谱法是一种有效的研究分子间作用的方法。当小分子与DNA作用时,化合物所处的环境受到DNA存在的影响,分子的紫外吸收峰会出现相应的特征变化(包括红移、蓝移或者增色、减色效应)[14,15]。
以Tris缓冲液1为参比,向4´ 10-5mol× L-1的UC溶液中滴加一定体积的DNA溶液,使得[DNA]/[UC]的比例不断增加,记录溶液在260nm处的吸光度值。表1中为不同[DNA]时,在260nm处混合体系的实际测得的吸光度值和将两者(DNA和UC)简单混合(彼此无相互作用的)的计算吸光度值(即相应浓度的DNA和UC各自单独的吸光度值之和)。
表
1 不同[DNA]浓度下混合体系的吸光度值(260nm)Table 1 the absorption of the mixture where UC (4×10-5mol·L-1) was mixed with different concentration of DNA
[DNA]´ 105 mol× L-1 |
0 |
0.975 |
1.949 |
2.924 |
3.899 |
5.848 |
7.797 |
9.947 |
11.730 |
实测吸光度值 |
0.23055 |
0.32807 |
0.41489 |
0.52342 |
0.61674 |
0.75269 |
0.88800 |
1.01902 |
1.15011 |
计算吸光度值 |
0.23055 |
0.29412 |
0.35768 |
0.42125 |
0.48481 |
0.61194 |
0.73908 |
0.86621 |
0.99334 |
| DA (实测-计算) |
0 |
0.03395 |
0.05721 |
0.10217 |
0.13193 |
0.14075 |
0.14892 |
0.15279 |
0.15677 |
根据上表作图(图2),从图中我们可以看出,保持UC浓度不变,当[DNA]/[UC]≤1时,随着DNA浓度的不断增大,混合体系的吸光度迅速增大,并且在[DNA]/[UC]=1时,DA /
AUC达到了57%,这说明DNA与UC之间确实发生了作用,并且生成的产物在测定波长260nm处的摩尔吸光系数比单纯的DNA或者UC的摩尔吸光系数都大。当[DNA]/[UC]> 1,随着[DNA]的增加,吸光度仍有一定的增加,但是比[DNA]/[UC]< 1时要平缓的多,基本趋平。以上结果说明,在此实验条件下,DNA与UC优先生成1:1的超分子化合物,这与以前的报道结果是相符的[13]。但是运用紫外的方法目前尚不足以确定在此情况下UC与DNA作用的具体位点及作用模式,有待于进一步深入的研究。
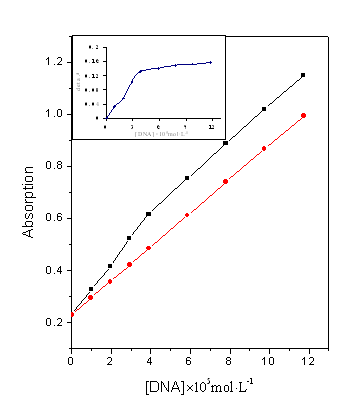
图2 不同[DNA]下体系吸光度的变化图(■代表实测吸光度值;●代表计算吸光度值); 插图为吸光度变化值对[DNA]的变化曲线
Fig.2 The variation
of the absorption of the mixture with increasing of the concentration of DNA(■denotes the absorption measured;●denotes the absorption
calculated); Inserted was the curve of DA
via [DNA]
3.2
粘度法研究UC与CT-DNA的相互作用粘度测试是检测小分子化合物与DNA结合模式的最有效的方法之一,其结果比光谱数据更具说服力[16,17]。当小分子化合物以静电、沟面结合等非插入方式与DNA作用时,DNA溶液的粘度无明显变化;当小分子化合物以经典插入方式与DNA作用时,DNA相邻碱基对的距离会因小分子的插入而变大,导致DNA双螺旋伸长,使DNA溶液的粘度增加,并且结合强度越大,粘度的变化越大;而当化合物与DNA以部分插入方式作用时,则可能使DNA双螺旋发生扭结,导致DNA溶液的粘度减小。
本节中,作者采用粘度的方法测试了UC对DNA粘度的影响,并与典型的以插入模式与DNA结合的配合物[Ru(bpy)2dppz](ClO4)2比较。
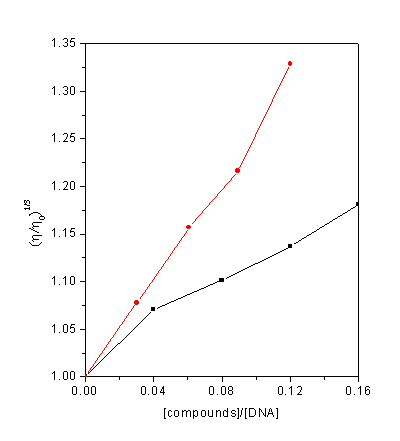
图3 不同浓度的UC对DNA溶液相对粘度的影响: ■ 代表不同浓度的[Ru(bpy)2dppz](ClO4)2对DNA粘度的影响;●代表不用浓度的UC对DNA粘度的影响
Fig.3 Effect of increasing amounts of UC and [Ru(bpy)2(dppz)]2+ on the relative viscosity of CT-DNA at 30±0.01oC. [DNA]=2.5×10-4mol·L-1
如图3所示,我们以经典的以插入模式结合DNA的配合物[Ru(bpy)2dppz](ClO4)2作为对照,随着UC浓度的不断增加,DNA溶液的粘度增大,这说明UC以插入的模式与DNA相结合,并且比[Ru(bpy)2dppz](ClO4)2的插入强度更大。我们推测这可能是UC上的碱基,即尿嘧啶作为插入基团,插入到了DNA的碱基对当中。这种超分子作用说明UC中的尿嘧啶基团确实起到了识别DNA的作用,是UC与DNA结合成超分子化合物的结合位点。当然,UC插入到DNA分子中的具体位置,即DNA的接纳位点尚需进一步的确认。
3.3 UC对DNA断裂作用的研究
琼脂糖凝胶电泳实验可以用来研究小分子化合物对DNA的断裂作用。在凝胶电泳中具有不同分子量的DNA的迁移速率是不同的,分子量越小,迁移速率越大。质粒DNA中存在三种构型。其中共价闭环的超螺旋Form I 的结构最为紧密,迁移速率最大,走在最前面。线形的Form III 走在其次。而开环缺刻的Form II 由于结构比较松散,走在最后面[18]。
当小分子化合物以插入模式与DNA作用时,往往会导致DNA分子的解旋和断裂,产生不同构型的DNA分子,在凝胶电泳中就会出现不同的条带。
在前面工作的基础上,作者首先研究了不同浓度的UC对DNA的作用,发现在只有UC和pBR322 DNA存在的条件下,37oC下恒温12小时,并没有发现pBR322 DNA发生断裂(电泳图此处未列出)。但是当向反应液中加入一定量的H2O2时,DNA发生了断裂。如图4所示,当只有DNA或DNA+H2O2存在时,DNA并没有发生断裂;而当向电泳反应液中加入UC后,DNA发生了断裂,从Form I向Form II转化,并且随着UC浓度的增加,有更多的Form I 型pBR322 DNA转变为Form II 型缺刻DNA。这说明了UC的存在,对H2O2断裂DNA起到了超分子催化作用,而这种催化作用来自于UC对DNA的分子识别,可以归属为一种基于分子识别的超分子催化作用[19]。这个结果为开发新型的含有杯芳烃基团的DNA定点切割模拟酶试剂提供了理论支持。

图4 不同UC浓度下pBR322 DNA的断裂情况;条件:37oC保温12小时;0 DNA;1 DNA+3m LH2O2;2 DNA+3mL H2O2 +10m LUC;3 DNA+3m LH2O2+20m LUC;4 DNA+3mL H2O2 +30m LUC;5 DNA+3mL H2O2+40mL UC;6 DNA+3mL H2O2 +50mL UC
Fig.4 Cleavage of pBR322 DNA in the presence of UC, kept at 37oC for 12 hours. Lane 0: DNA alone; 1 DNA+3mL H2O2;2 DNA+3mL H2O2 +10mL UC;3 DNA+3mL H2O2+20mL UC;4 DNA+3mL H2O2 +30mL UC;5 DNA+3mL H2O2+40mL UC;6 DNA+3mL H2O2 +50mL UC
4 小结
本文采用紫外滴定和粘度测试的方法研究了新型杯芳烃衍生物UC与CT-DNA的相互作用,结果发现:UC可以以经典插入的模式与DNA作用,并与DNA形成1:1的超分子化合物;我们推测可能是UC中的碱基尿嘧啶插入到了DNA的碱基对中间,起到了识别DNA的作用。同时,我们采用琼脂糖凝胶电泳的方法,研究了pBR322 DNA的断裂情况,结果发现:在温度37oC条件下,UC的存在对H2O2断裂pBR322 DNA起到了超分子催化的作用,使DNA从闭环超螺旋的Form I 型转变为缺刻的Form II型。本文为开发新型的含有杯芳烃基团的用于DNA定点切割的模拟酶试剂提供了理论的支持。
REFERENCES
[1] Gutsche C. D., Dhawan B., No K. H., et al, J. Am. Chem. Soc., 1981, 103: 3782
[2] RondelezY., Bertho G., and Reinaud O., Angew. Chem. Int. Ed., 2002, 41, 1044
[3] Cao Y. D., Zheng Q. Y., Chen C. F., Hu H. M., Huang Z. T., Inorg. Chim. Acta, 2004,
357: 316
[4] Rondelez Y., Duprat A., and Reinaud O., J. Am. Chem. Soc., 2002, 124: 1334
[5] Seneque O., Rager M. N., Giorgi M.,and Reinaud O., J. Am. Chem. Soc., 2000, 122: 6183
[6] Sénèque O., Rager M. N., Giorgi M.,et al, J. Am. Chem. Soc. 2001,
123: 8442
[7] Goto K., Murakami Y., Ueoka R., J. Mol. Catal. B: Enzymatic, 2001, 11: 985
[8] Molenveld P., Engbersen J. F. J., Kooijman H., J. Am. Chem. Soc. 1998, 120:
6726
[9] Rondelez Y., Bertho G., Reinaud O., Angew. Chem. Int. Ed. 2002, 41(6): 1044
[10] Sénèque O., Rager M. N., Giorgi M., et al, J. Am. Chem. Soc. 2005, 127:
14833
[11] Bukhaltsev E., Frish L., Cohen Y., et al, Org. Lett., 2005, 7(23): 5123
[12] Ikeda A., Ejima A., Nishiguchi K., et al, Chemistry Letters, 2005, 34(3): 308
[13] Shi H. J., Shi X. F., Yao T.M., et al, Chinese Journal of Chemistry, in press
[14] Baudoin O., Gonnet F., Lehn J. M., et al, Chem. Eur. J. 1999, 5(9): 2762
[15] He Y.B., Xiao Y. J., Meng L. Z., et al, Tetrahedron Letters, 2002, 43: 6249
[16] Satyanarayana S., Dabroniak J.C., Chaires J.B., Biochemistry, 1992, 31: 9319
[17] Satyanarayana S., Dabroniak J.C., Chaires J.B., Biochemistry, 1993, 32: 2573
[18] Barton J. K., Raphael A. L., J. Am. Chem. Soc., 1984, 106: 2466
[19] Lehn J. M., Nobel lecture, Dec. 8, 1987: 462